Descarga y visualiza datos de casos COVID-19 en R
En este tutorial, te enseñamos a descargar datos de los casos confirmados del 19-nCoV del European Center for Disease Control (ECDC) para su análisis y visualización en R. Este tutorial contiene los siguientes pasos:
- Prepara el environment: librerías
- Descarga los datos
- Pre-procesa los datos
- Selecciona los países de tu interés
- Visualiza los datos con ggplot
- Guarda el grafico con ggsave()
Prepara el environment: (des)carga las librerías
Si no has instalado R Studio, descárgalo aquí. Aunque puedes usar otro IDE, aquí un resumen de por qué recomendamos R Studio. Para (des)cargar las librerías necesarias, corre el siguiente código:
# Para instalar
install.packages(c("dplyr", "ggplot2", "ggrepel", "zoo"))
# Para cargarlos al environment
library(dplyr) # para manipular datos
library(ggplot2) # gráficos
library(ggrepel) # etiquetas gráficos
library(zoo) # fechas
Descarga los datos
Los datos globales de casos del coronavirus están disponibles aquí. Para descargarlos y almacenarlos en un data frame:
# Descarga los datos
data<-read.csv("https://opendata.ecdc.europa.eu/covid19/casedistribution/csv")
# Explora el formato de los datos
str(data) # estructura
head(data) # primeras 10 filas
Pre-procesa los datos
Para facilitar la visualizacion de los datos, vamos a cambiar el formato de la columna dateRep (o fecha de reporte) al tipo Date con el paquete zoo. Además, creamos la columna dsfc, o “days since first case”, que especifica cuántos días transcurrieron entre el primer caso reportado y cada nuevo reporte, por país. También creamos la variabe totalCases con el total de casos acumulados.
# Convierte la fecha a formato Date
data<-data%>%mutate(date=as.Date(dateRep, '%d/%m/%Y'))
# Calcula dias desde primer caso reportado (variable dsfc)
data<-data%>%arrange(countriesAndTerritories, desc(date))%>%filter(cases>0)%>%
group_by(countriesAndTerritories)%>%mutate(dsfc=date-min(date))
# Calcula casos reportados cumulativos
data<-data%>%mutate(totalCases=NA)
for(i in (dim(data)[1]):1){
if(i==dim(data)[1]){
data$totalCases[dim(data)[1]]<-data$totalCases[dim(data)[1]]
}
if(i!=dim(data)[1]){
if(data$countriesAndTerritories[i+1]==data$countriesAndTerritories[i]){
data$totalCases[i]=(data$totalCases[i+1]+data$cases[i])
}
if(data$countriesAndTerritories[i+1]!=data$countriesAndTerritories[i]){
data$totalCases[i]=data$cases[i]
}
}
}
Selecciona los países de tu interés
Haz un vector con los nombre de los territorios de tu interés. Acá seleccionamos algunos países de Latinoamérica:
# Nombres de territorios para visualizar
data$countriesAndTerritories%>%unique() # Ver nombres únicos de territorios
la<-c("Argentina", "Bolivia", "Brazil", "Mexico", "Peru", "Uruguay", "Dominican_Republic")
Visualiza los datos con ggplot
Se ha generado un debate interesante en cuanto a la forma “correcta” de visualizar datos de casos confirmados del COVID-19 (ver hilo en Twitter). Acá graficamos 3 :
A) Casos acumulados por millón de habitantes (eje y) VS Días transcurridos desde el 1 caso fue reportado (eje x)
B) Casos acumulados por millón de habitantes (eje y) VS Días transcurridos desde que 1 caso por millón de habitantes fue reportado (eje x)
C) Logaritmo de casos acumulados por millón de habitantes (eje y) VS Días transcurritos desde 1 caso por millón de habitantes fue reportado (eje x).
El siguiente código
- Filtra
datapara los paises en el vectorla - Grafica líneas y puntos con
geom_lineygeom_point - Asigna un color a cada país
aes(color=countriesAndTerritories) - Modifica etiqueta de los ejes y la leyenda con
xlab,ylabylabs(color=Pais) - Agrega etiquetas con el nombre del país al último día de reporte, minimizando la superposición con
geom_label_repel
# A- Visualizar casos acumulados desde el primer caso reportado por millon de habitantes
ggplot(data%>%filter(countriesAndTerritories%in%la)) +
geom_line(aes(x=dsfc, y=totalCases/(popData2018/1000000), color=countriesAndTerritories)) +
geom_point(aes(x=dsfc, y=totalCases/(popData2018/1000000), color=countriesAndTerritories)) +
xlab("Dias desde el primer caso confirmado") +
ylab("Casos acumulados por millón de habitantes") +
labs(color="Pais") +
geom_label_repel(data=data%>%filter(countriesAndTerritories%in%la)%>%
group_by(countriesAndTerritories)%>%
summarize(dsfcmax=max(dsfc), totalCasesmax=max(totalCases),
popData2018=max(popData2018)),
aes(x=dsfcmax, y=totalCasesmax/(popData2018/1000000) ,
label=countriesAndTerritories, color=countriesAndTerritories),
size=2, show.legend = F) +
theme_bw()
# B- Visualizar casos acumulados desde el primer caso reportado por millon de habitantes
# Calcular días desde 1 caso/millon habitantes reportado
data1<-data%>%mutate(popMillon = popData2018/1000000)%>% filter(totalCases>=popMillon)%>%
group_by(countriesAndTerritories)%>%mutate(dscpm=date-min(date))
ggplot(data1%>%filter(countriesAndTerritories%in%la)) +
geom_line(aes(x=dscpm, y=(totalCases/(popData2018/1000000)), color=countriesAndTerritories)) +
geom_point(aes(x=dscpm, y=(totalCases/(popData2018/1000000)), color=countriesAndTerritories)) +
xlab("Días desde reporte de 1 caso/millón de habitantes") +
ylab("Casos acumulados por millón de habitantes") +
labs(color="País") +
geom_label_repel(data=data1%>%filter(countriesAndTerritories%in%la)%>%
group_by(countriesAndTerritories)%>%
summarize(dscpmmax=max(dscpm), totalCasesmax=max(totalCases),
popData2018=max(popData2018)),
aes(x=dscpmmax, y=(totalCasesmax/(popData2018/1000000)) ,
label=countriesAndTerritories, color=countriesAndTerritories),
size=2, show.legend = F) +
theme_bw()
# C- Visualizar log de casos acumulados desde el primer caso reportado por millon de habitantes
ggplot(data1%>%filter(countriesAndTerritories%in%la)) +
geom_line(aes(x=dscpm, y=log(totalCases/(popData2018/1000000)), color=countriesAndTerritories)) +
geom_point(aes(x=dscpm, y=log(totalCases/(popData2018/1000000)), color=countriesAndTerritories)) +
xlab("Días desde reporte de 1 caso/millón de habitantes") +
ylab("Log(Casos acumulados por millón de habitantes)") +
labs(color="País") +
geom_label_repel(data=data1%>%filter(countriesAndTerritories%in%la)%>%
group_by(countriesAndTerritories)%>%
summarize(dscpmmax=max(dscpm), totalCasesmax=max(totalCases),
popData2018=max(popData2018)),
aes(x=dscpmmax, y=log(totalCasesmax/(popData2018/1000000)) ,
label=countriesAndTerritories, color=countriesAndTerritories),
size=2, show.legend = F) +
theme_bw()
Figura A
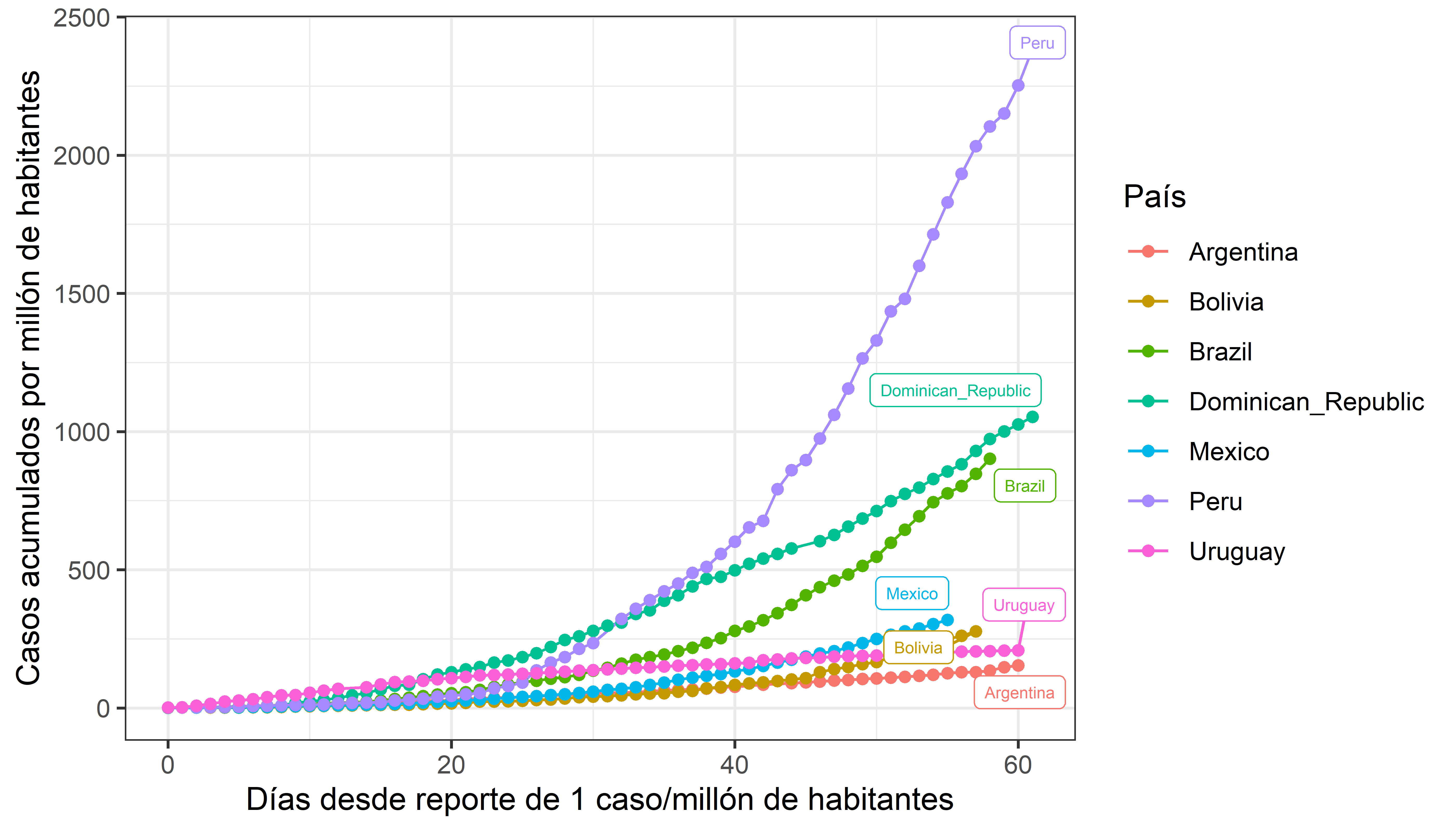
Este gráfico es similar a los que circularon al principio de la pandemia. Sin embargo, puede desfavorecer a países con mayor población (por ejemplo, Perú). Carl T. Bergstrom propuso entonces graficar en el eje X desde el momento en el que un % específico de la población de cada país se contagiara (ver figura B).
Figura B
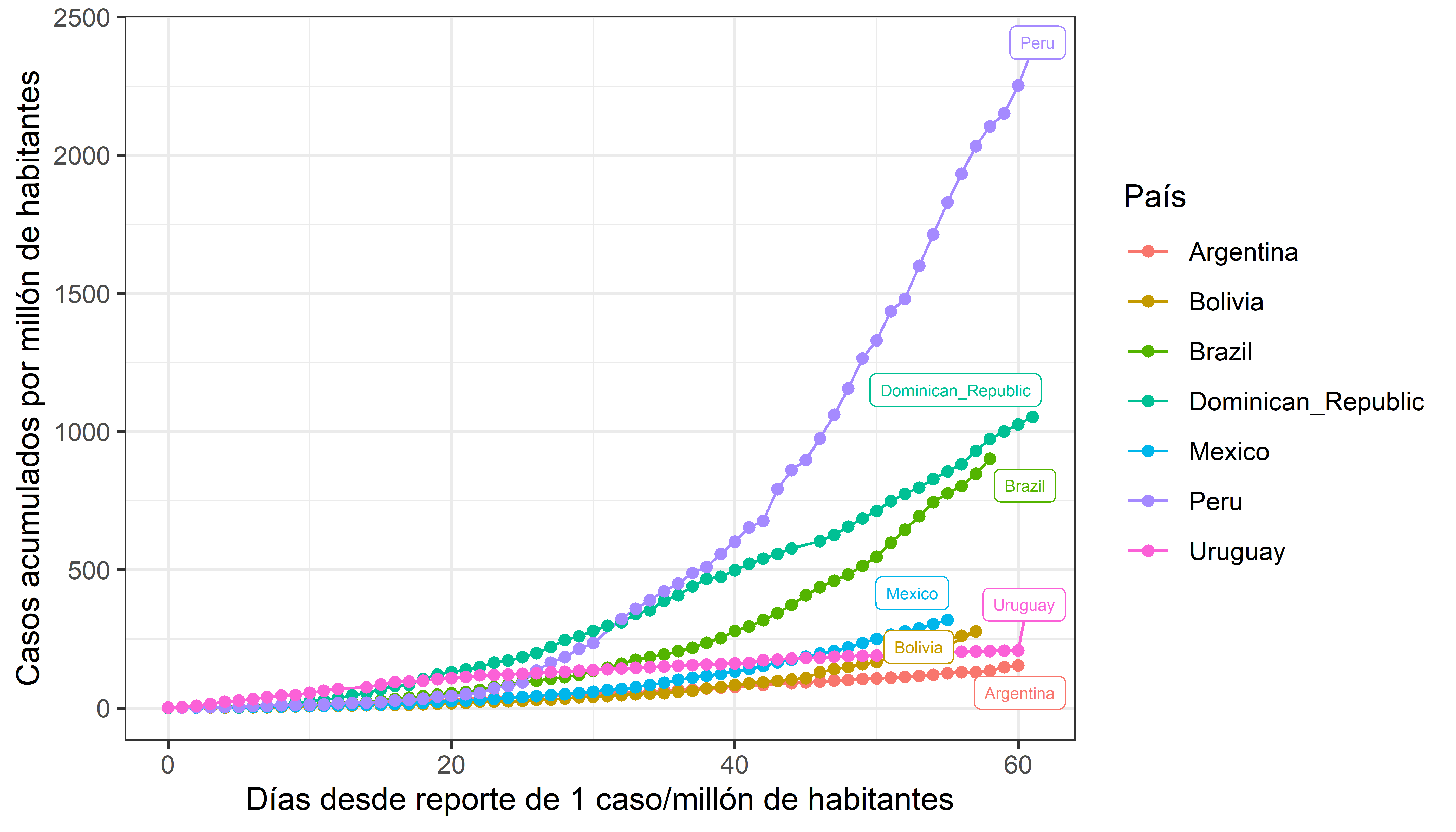 En este gráfico, el eje x indica el número de días que han pasado desde que el 0.0001% de la población fue contagiado (1 caso por cada 1,000,000 de habitantes); ajusta este porcentaje como te convenga en el código. El crecimiento cuasi exponencial de los casos en este gráfico puede ser difícil de interpretar (ver figura C).
En este gráfico, el eje x indica el número de días que han pasado desde que el 0.0001% de la población fue contagiado (1 caso por cada 1,000,000 de habitantes); ajusta este porcentaje como te convenga en el código. El crecimiento cuasi exponencial de los casos en este gráfico puede ser difícil de interpretar (ver figura C).
Figura C
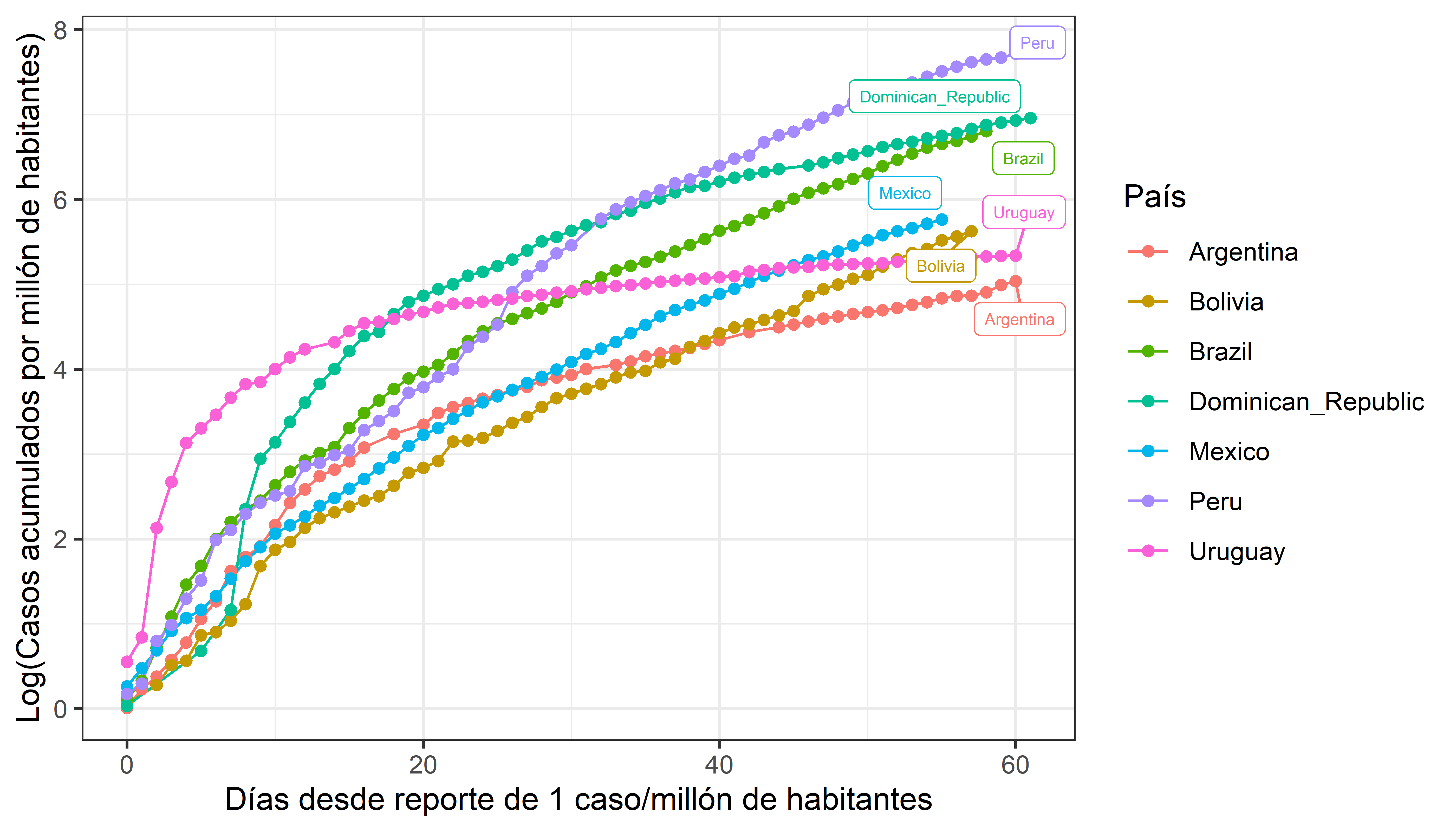
En este gráfico, la pendiente refleja la tasa de crecimiento de casos, la altura refleja la prevalencia y el tiempo relativo de la figura B se mantiene. Fuente.
Guarda el gráfico con ggsave()
Este código guarda el gráfico en display en formato png, en el path folder/ejemplo y con nombre de archivo covid19-casos. Para cambiar el formato, sustituye png por un valor de c("jpeg", "tiff""eps", "ps", "tex", "pdf", "jpeg", "tiff", "png", "bmp", "svg" o "wmf")
# Guarda el gráfico en display con ggsave()
ggsave("folder/ejemplo/covid19-casos.png",
device="png", dpi=800,
width=unit(7, "in"), height=unit(4 ,"in"))
Ver la versión en Inglés de este tutorial.
Si tienes preguntas, puedes contactarme.